
在调查犯罪时,法医科学家有时会使用DNA证据来识别嫌疑犯。但是,DNA不仅识别信息,还包括我们基因组成的线索。DNA分离用于仔细观察DNA链,但传统方法很耗时。为了加快DNA分离过程,密苏里科学技术大学的研究人员转向了ComsolMultiphysics®软件。
仔细观察我们的基因组成
DNA的分子结构是复杂的:它是由长核苷酸链组成的双螺旋聚合物。通过将样品分解为不同大小的片段,研究DNA要容易得多。
核苷酸或碱基对DNA是鸟嘌呤(G),腺嘌呤(A),胸腺嘧啶(T)和胞嘧啶(C)。研究人员试图在基因组测序和医学诊断等领域中理解这些遗传“字母”的序列定位基因看看它们如何在生物体中一起工作。如果没有DNA分离,这项工作就不容易做到 - 毕竟,人类基因组已经结束30亿个DNA碱基对呢
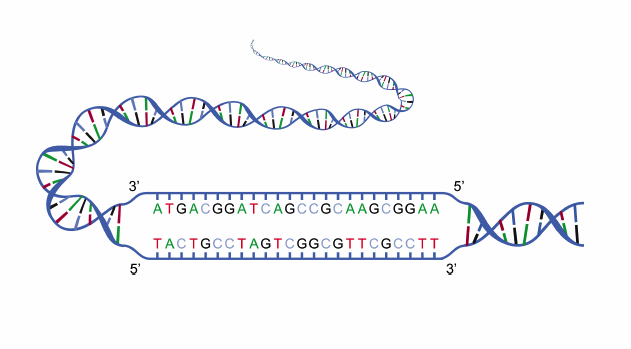
包含基对字母g,a,t和C的DNA链的插图Wikimedia Commons。
DNA分析的其他示例最近也进入了聚光灯。您可能会熟悉邮寄DNA测试套件,以帮助您了解有关祖先的更多信息。当基因测试公司将DNA样品数字化时,它看起来像是核苷酸G,A,T和C字母的一条长链。这些公司使用算法将基因组中的DNA与参考数据集进行比较。然后,该算法确定您的DNA样本与每个参考设置的匹配程度如何,以查看您最有可能属于的祖先组。该算法仅与参考集一样好,因此与数据库中的其他算法相比,某些祖先组的代表性不足。
在法医等领域,DNA分析有助于科学家比较遗传物质的样本。由于很少有两个人具有相同的DNA模式,因此法医科学家可以将DNA分子切片中的模式与参考数据库进行比较,例如由美国FBI管理的组合DNA指数系统(Codis)。但是,像Codis这样的系统仅限于它们所包含的DNA轮廓。研究人员开始使用如上所述的祖先数据库通过称为家族性DNA的概念来扩展其搜索。例如,2018年,警方调查了金州杀手的案件犯罪现场DNA反对家谱现场数据库并找到了与遥远亲戚的部分匹配。最终,这有助于他们缩小搜索范围并确定所谓的嫌疑人。
研究具有DNA分离的核苷酸链中的碎片链接
一种用于分离DNA分子(主要用于取证)的常见技术是凝胶电泳,它涉及通过凝胶迁移带负电荷的核酸分子的迁移。当施加电流时,较小的分子在凝胶上移动的速度比较大的分子移动,因此片段根据尺寸分离为带。为了可视化这种分离,使用放射性染料。
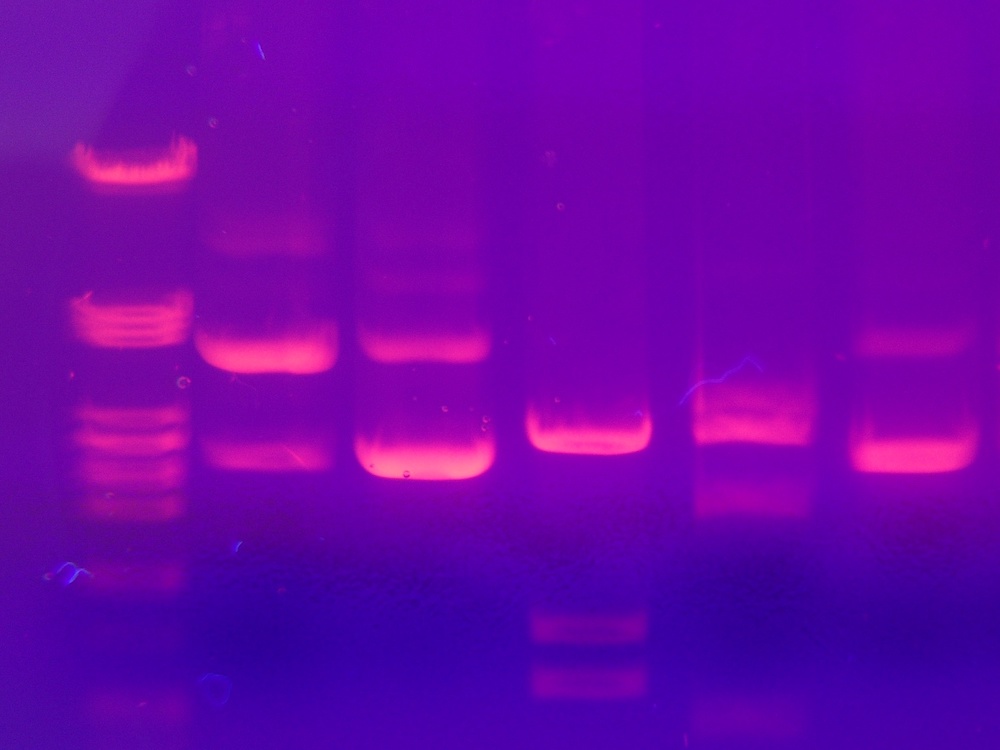
凝胶电泳结果的一个例子。Mnolf的图像 - 自己的作品。获得许可CC BY-SA 3.0, 通过Wikimedia Commons。
还有另一种方法可以在不使用凝胶或电场的情况下更有效地将长长的DNA链分开:熵诱捕。在这个基于微芯片的系统中,建立了不同高度的熵陷阱阵列(结构化微通道),因此狭窄的通道间隙比DNA分子的回旋直径小得多。分子可以根据链长度分离。当带负电荷的DNA分子通过这些通道驱动时电泳力,洗脱时间取决于长度。DNA分子越长,将其吸入小通道的可能性就越大,因为较长的分子占据了更多的表面积。

一系列熵陷阱的示意图,宽通道中的DNA分子流入狭窄的通道。图像由密苏里科学技术大学提供。
尽管熵诱捕比其他分离方法更快,更高效,但所需设备的设计和制造需要大量时间,并且可能会昂贵,因为它依赖于试用和错误。自从发现熵诱捕方法以来,研究人员已经进行了计算研究以优化设计并研究这些设备内的分离机制,但是尚未使用商业软件来模拟这些熵陷阱系统……直到现在。
使用comsolMultiphysics®在熵陷阱系统中模拟聚合物动力学
为了找出他们是否可以通过市售模拟软件节省时间,密苏里科学技术大学的研究人员使用COMSOLMultiphysics®建立了其熵陷阱系统和聚合物动力学模拟,并将其结果与实验数据进行了比较。
该研究团队由Joontaek Park,James Jones,Meyyamai Palaniappan,Saman Monjezi和Behrouz Behdani组成,他说:“微通道模拟中的DNA动力学是具有挑战性的,因为两种不同的模拟 - 复杂的微流体和Polymetry和Polymemer和Polymer Molecer和Polymer Molecer Motemer和Polymer Molecer Motemer和Polymer Molecer Motemer和Polymer Meterics的现场计算 -合并。”幸运的是,他们补充说:“Comsol®可以相对容易处理以前的模拟”,并且“Comsol®可以在DNA或单聚合物分子模拟区域中打开新页面。”
使用附加组件粒子跟踪模块,团队表演布朗动力学DNA链的模拟。该链是在牛顿流体中作为单聚合物的珠链模型设置的CFD模块。至于珠子本身,它们被视为布朗颗粒,以解释链条穿过周围溶剂时的随机运动。
为了描述每个珠之间的弹簧力,他们使用了另一个众所周知的模型,即蠕虫样链(WLC),该模型描述了半串联聚合物的行为。在WLC之外,研究团队还利用Lennard -Jones的潜力来防止珠子相互穿透。设置熵阵列几何形状(如下所示)之后Hs比典型的DNA分子的回旋直径小得多,研究人员使用了AC/DC模块在整个通道上创建电势场。
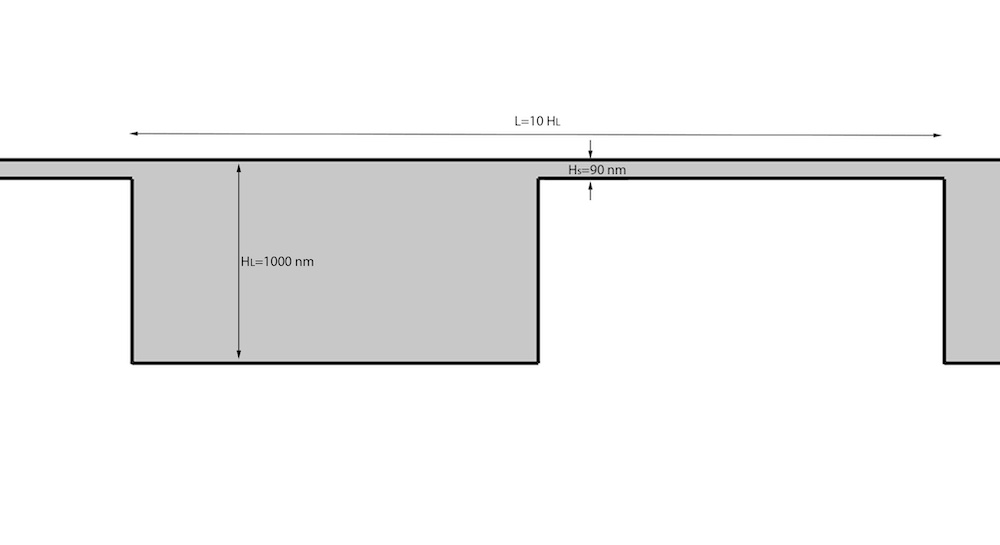
模拟中使用的通道结构的示意图。图像由密苏里科学技术大学提供。
评估模拟结果
研究团队使用有限元方法。在这里可以看到电场方向,箭头还指示了DNA分子运动的方向。
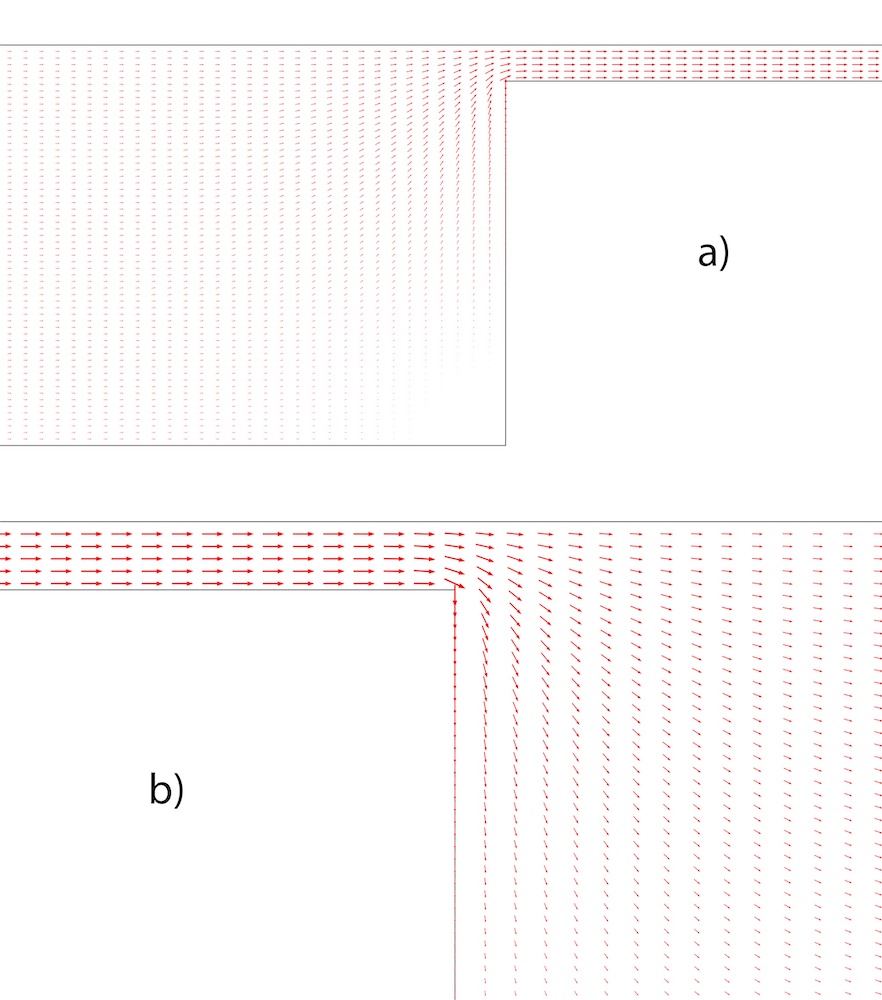
右角(a)和宽通道的左角(b)的电场通量向量。图像由密苏里科学技术大学提供。
接下来,研究人员根据长度模拟了DNA分子的质量轨迹nb= 2、4和16珠长度,因为它们定期流入狭窄的通道。下面显示在相同距离的每个分子的轨迹。正如从上方的电场矢量所预期的那样,分子在狭窄的通道中移动越快,分子越长(珠子越多),移动的速度就越快。同时,较短的分子沿其轨迹的速度降低,而DNA分子的分布表明了更具扩散的模式,从而通过将它们远离电场最强的区域来降低通过通道的总体速度。
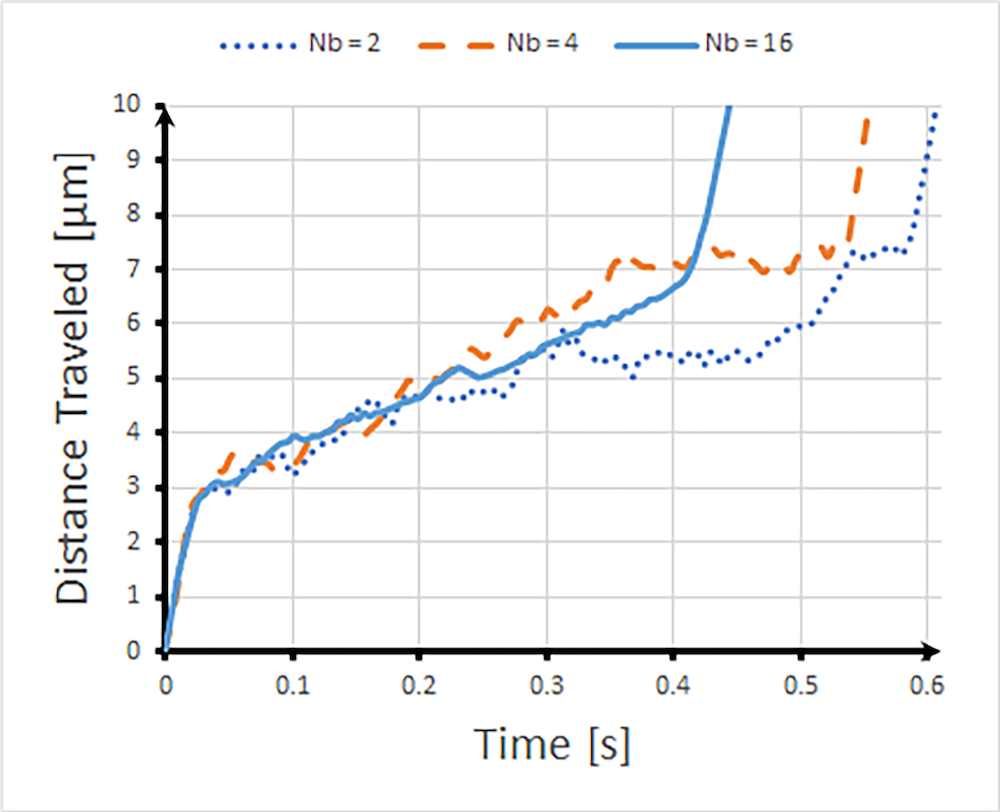
DNA分子的质量轨迹nb= 2、4和16。图像由密苏里州科学技术大学提供。
可以在下面的动画中看到此比较的短珠长度,nb= 2,中珠长度nb= 4,长珠长度,nb= 16。如传说上的单元所示,动画中的颜色在任何时间点显示了粒子的速度。如预期的那样,DNA分子的表面越大,将其拖入较小的通道的可能性就越大。(请注意,动画比实时慢约10倍。如果您希望进一步降低速度,则可以悬停在动画上,然后单击齿轮图标。)
nb= 2较短的DNA分子在熵陷阱通道中流入和流出宽通道。动画提供密苏里科学技术大学。
nb= 4中间长度DNA分子在熵陷阱通道中流入和流出宽通道。动画提供密苏里科学技术大学。
nb= 16个更长的分子在熵陷阱通道中流入和流出宽通道。动画提供密苏里科学技术大学。
研究人员能够确认他们的仿真结果与熵陷阱中DNA链的轨迹的实验数据非常吻合,这些结果表明,较长的DNA链确实比较短的链更快地洗脱了。
使用COMSOL多物理用于聚合物动力学仿真为进一步研究开辟了可能性,因为这是使用市售软件进行此类仿真的第一个试验。该团队说,“ COMSOL多物理学是一种非常流行且用户友好的模拟工具”,此外,将软件用于聚合物动力学的扩展将“增强相关的应用和仿真研究”。
至于他们自己的未来研究?该团队补充说,他们可以看到对惯性效应,聚合物构型(分支聚合物)效应和DNA-碳纳米管相互作用进行研究。
下一步
有关密苏里科学技术大学研究人员工作的更多详细信息,请单击下面的按钮:
参考
- S. Monjezi,B。Behdani,硕士Palaniappan,J.D。Jones和J. Park,“微型制造设备中DNA分离的计算研究:一般方法和最新应用的综述”,,ADV。在化学中。工程。科学。,7(4),第362–393页,2017年。




评论(0)